Taxonomische Identifikation der Unsichtbaren
Woher wissen wir, welche Pilze und Bakterien Pflanzen besiedeln oder wie viele verschiedene Bakterienarten auf einem Pflanzenblatt zu finden sind? Traditionell nutzt man die klassische Methodik der Spezies-Identifizierung von Tieren oder Pflanzen, die vor allem Merkmale der äußeren Form und Gestalt wie z. B. Blätterform und -anordnung, Form und Farbe der Schoten und Früchte sowie Blütentypus und -farbe bei Pflanzen heranzieht (Abbildung 1).
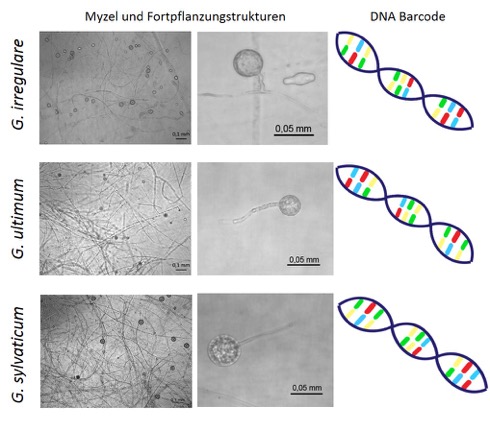
Diese Methodik ist schwer anzuwenden auf Organismen, die mit dem nackten Auge nicht erkennbar sind und oft unter einem Mikroskop nicht von Artgenossen zu unterscheiden sind. Meist sind die äußeren Merkmale eng verwandter Arten dieser Organismen sehr ähnlich und dienen kaum zur Spezies-Identifizierung (Abbildung 2). Allerdings variieren diese Mikroben in ihrer Erbsubstanz (DNA), was wir nutzen können, um die verschiedenen Arten zu detektieren.
DNA Barcoding, die Identifikation einer Spezies mittels ihrer Erbsubstanz, gibt es seit Jahrzehnten. Spezifische, für die Artenzuordnung geeignete, genetische Marker wurden für verschiedene Organismengruppen identifiziert und die Reihenfolge der Erbsubstanzbestandteile (Sequenzen) dieser Markerregion von bekannten Organismen erhoben, welche man DNA-Barcodes nennt (Abbildung 3). Nicht jede Stelle in der Erbsubstanz ist gleich informativ, daher wurden für verschiedene Organismengruppen die informativsten Stellen identifiziert. Diese sind z.B. ribosomale RNA im Falle von Bakterien und Protisten oder ein Gen innerhalb der Atmungskette (Cytochrom Oxidase I kodierend) für viele Tiere z. B. Insekten. Mit bekannten Sequenzen können nicht nur bekannte Arten in einer Probe identifiziert, sondern auch bisher unbekannte Verwandte entdeckt werden.
DNA Barcoding einer Gemeinschaft von verschiedenen Spezies bezeichnet man als Metabarcoding. Metabarcoding ist essentiell, um komplexe mikrobielle Gemeinschaften zu untersuchen, zum Beispiel solche, die mit Pflanzen zusammenleben. Obwohl wir schon viele Kenntnisse über Pflanzensymbionten und -pathogene haben, kennen wir immernoch nicht die Gesamtheit aller mit Pflanzen interagierender Mikroben. Diese wollen wir identifizieren, um besser die vielfältigen Interaktionen zwischen Wirtspflanzen und Mikroben untersuchen zu können.
Die Identifizierung von Oomyzeten und anderen Protisten assoziiert mit Kreuzblütlern ermöglicht, dass wir spezifische Interaktionen zwischen diesen Organismen, ihren Wirtspflanzen und anderen an der Wirtspflanze lebenden Mikroorganismen im Detail untersuchen können. Langfristig ist unser Ziel, mikrobielle Populationen zum Nutzen der Pflanze zu beeinflussen.
Planter’s Punch
Unter der Rubrik Planter’s Punch wird jeden Monat ein bestimmter Aspekt des CEPLAS Forschungsprogramms vorgestellt. Alle Beiträge werden von Mitgliedern der Graduiertenschule und des Postdoc Programms erstellt.
Zum Nachlesen
www.barcodeoflife.org
www.qbol.org/en/qbol.htm
Cristescu, M.E. (2014) From barcoding single individuals to metabarcoding biological communities: Towards an integrative approach to the study of global biodiversity. Trends Ecol Evol 29: 566–571. [Abstract]
Hebert, P.D.N., Cywinska, A., Ball, S.L., and deWaard, J.R. (2003) Biological identifications through DNA barcodes. Proc R Soc B Biol Sci 270: 313–321. [Abstract]
Hajibabaei, M., Singer, G.A.C., Hebert, P.D.N., and Hickey, D.A. (2007) DNA barcoding: how it complements taxonomy, molecular phylogenetics and population genetics. Trends Genet 23: 167–172. [Abstract]