Forward genetics – Eine effiziente Screening-Technik um neue Gene zu entdecken, die die C4 Blattanatomie ermöglichen
Pflanzen die C4 Photosynthese betreiben sind effizienter im Umgang mit CO2, Stickstoff- und Wasserhaushalt und können daher unwirtlichen Bedingungen wie hoher Lichteinstrahlung, hohen Temperaturen und Trockenheit besser widerstehen. Einer der Hauptgründe für diesen Anpassungsvorteil ist die Evolution der C4-spezifischen Kranz-Blattanatomie. Pflanzenblätter mit Kranz-Anatomie haben viele Blattvenen eng nebeneinander, die von vergrößerten Bündelscheidezellen mit sehr vielen Chloroplasten umgeben sind. Leider ist das regulative Gen-Netzwerk, das für die Entwicklung der Kranz-Anatomie verantwortlich ist, noch größtenteils unbekannt.
Forward genetics ist eine großformatiger Screening-Ansatz, um die genetische Basis dieses Phänotyps zu finden. Der Ausgangspunkt dieser Massenuntersuchung ist die Einführung von zufälligen Mutationen in das Pflanzengenom. Wir führen zwei unterschiedliche forward genetics Versuche gleichzeitig durch: Der Erste benutzt das das sogenannte Activation tagging (zu deutsch etwa: Identifizieren durch aktivieren), der Zweite ist Mutagenese mit EMS. Activation tagging ist nichts anderes als die Überexpression von nahgelegenen Genen durch die zufällige Insertion einer T-DNA Region, die einen starken Promotor beinhaltet. EMS-Mutagenese benutzt das alkylierende EMS (Ethylmethansulfonat) um einzelne Nukleotidaustausche im Genom zu erzeugen. Dies generiert meist Mutationen, die das jeweilige Gen unterbrechen und so einen Funktionsverlust bewirken.
Für unsere Großversuche benutzen wir Arabidopsis thaliana (dt. Ackerschmalwand oder auch Schotenkresse), eine gut untersuchte Modellpflanze, die C3 Photosynthese betreibt und einen basalen Bauplan der Kranz-Anatomie hat. Die Bündelscheidezellen um die Blattvenen sind jedoch klein und haben nur wenige Chloroplasten. Um nun in dem Screening leicht die Arabidopsis-Pflanzen erkennen zu können, die eine veränderte Blattanatomie haben, sind die Chloroplasten der Bündelscheidezellen mit dem grün fluoreszierenden Protein GFP markiert. Diese GFP-markierte Pflanzenlinie wurde dann entweder dem Activation tagging oder der EMS-Mutagenese unterzogen.
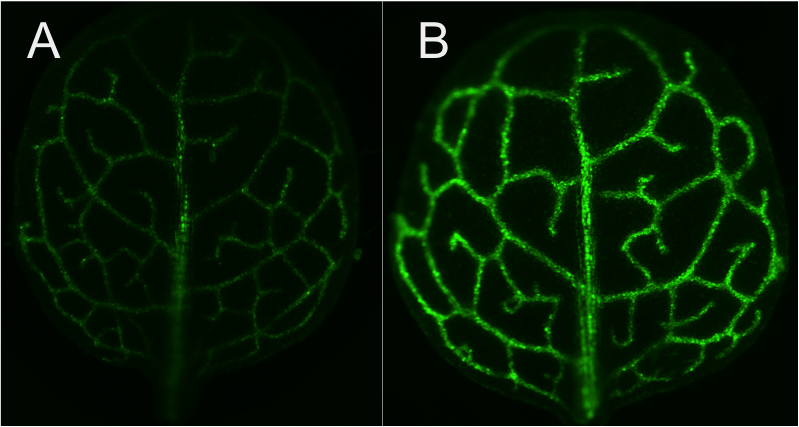
Da jeder Chloroplast in den Bündelscheidezellen der Pflanze mit GFP markiert ist, können wir annehmen, dass die Intensität der Fluoreszenz mit der Chloroplasten-Anzahl und davon abgeleitet auch mit der Größe der Bündelscheidezellen korreliert. Wir untersuchen daher von jeder einzelnen Pflanze ein Blatt, das wir abschneiden und dessen GFP-Fluoreszenz mit Hilfe eines speziellen Mikroskops messen. Wir suchen Pflanzen mit höherer oder geringerer Fluoreszenzintensität als unsere Referenzline (siehe Abbildung). Später verifizieren wir die Größe der Bündelscheidezellen und die enthaltene Chloroplastenzahl und isolieren die betroffenen Gene mit Hilfe von inverser PCR und Kartierung durch Sequenzierungs-Methoden. Die interessanten Kandidaten der identifizierten Gene werden dann in Kulturpflanzen wie Reis weiteruntersucht.
Die wichtigen Kulturpflanzen Reis, Weizen, Gerste oder Sojabohne sind alle Pflanzen, die C3 Photosynthese betreiben – wie Arabidopsis thaliana. Um ihre photosynthetische Effizienz unter den sich verändernden Klimabedingungen zu verbessern, können wir einen C4-Photosynthese-Weg implementieren; dafür ist das Verständnis der Kranz-Anatomie Grundvoraussetzung.
Kumari Billakurthi, Entwicklungs- und Molekularbiologie der Pflanzen, Heinrich Heine Universität Düsseldorf, Übersetzung von Meike Hüdig, Molekulare Physiologie und Biotechnologie der Pflanzen, Heinrich Heine Universität Düsseldorf
Planter’s Punch
Unter der Rubrik Planter’s Punch wird jeden Monat ein bestimmter Aspekt des CEPLAS Forschungsprogramms vorgestellt. Alle Beiträge werden von Mitgliedern der Graduiertenschule und des Postdoc Programms erstellt.
Zugehörige Publikation
Gowik U, Westhoff P (2011) The Path from C3 to C4 Photosynthesis. Plant physiology 155(1):56-63. [Abstract]